Introduction :
Cet article s’inspire du travail effectué par Louis Deharveng et David Porco (respectivement rattachés au Museum National d’Histoire Naturelle de Paris et de Luxembourg) qui exploitent l’outil génétique et qui ont eu l’amabilité de me faire parvenir des documents relatifs à leurs travaux sur les collemboles. L’accès à ces derniers nécessite des connaissances que ne possède pas forcément le néophyte, c’est pourquoi mon approche sur l’objet et la nature du « barcoding » reste très sommaire.
Généralités sur la cellule :
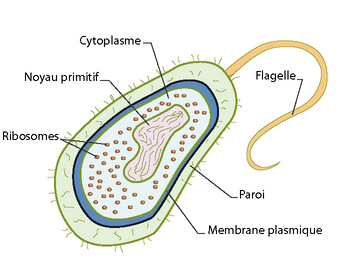 Il existe deux grands groupes de cellules, les cellules eucaryotes et les procaryotes. Les procaryotes (ci-contre) sont des bactéries ou des archées qui possèdent une paroi cellulaire et, dans la plupart des cas, un ADN circulaire (fermé, sans extrémité libre) mais qui ne possèdent pas de noyau, ce qui les distingue des cellules procaryotes. Ces dernières sont celles qui constituent le corps de l’homme et, plus largement, les organismes des quatre grands règnes du vivant (Animal, Plantes, Champignons, Bactéries). Regardons les d’un peu plus près.
Il existe deux grands groupes de cellules, les cellules eucaryotes et les procaryotes. Les procaryotes (ci-contre) sont des bactéries ou des archées qui possèdent une paroi cellulaire et, dans la plupart des cas, un ADN circulaire (fermé, sans extrémité libre) mais qui ne possèdent pas de noyau, ce qui les distingue des cellules procaryotes. Ces dernières sont celles qui constituent le corps de l’homme et, plus largement, les organismes des quatre grands règnes du vivant (Animal, Plantes, Champignons, Bactéries). Regardons les d’un peu plus près.
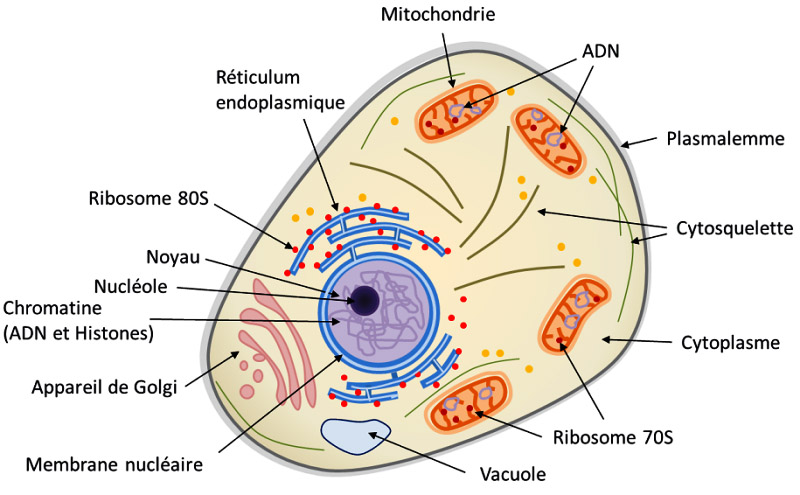 En plus du noyau, elle se composent de structures (organites) qui jouent des rôles importants. La membrane plasmique (plasmalemme) enveloppe le cytoplasme (constitué à 80 % d’eau) et délimite la cellule de son environnement. Le noyau stocke et protège l’essentiel de l’ADN. En son centre se trouve une zone dense (nucléole), siège de la transcription des ARNr (ARN Ribosomique) entrant dans la fabrication des ribosomes.
En plus du noyau, elle se composent de structures (organites) qui jouent des rôles importants. La membrane plasmique (plasmalemme) enveloppe le cytoplasme (constitué à 80 % d’eau) et délimite la cellule de son environnement. Le noyau stocke et protège l’essentiel de l’ADN. En son centre se trouve une zone dense (nucléole), siège de la transcription des ARNr (ARN Ribosomique) entrant dans la fabrication des ribosomes.
L’ARN (acide ribonucléique) est une molécule synthétisée dans la cellule à partir d’une matrice d’ADN. Elle correspond à une sorte de copie de ce dernier servant à véhiculer l’information dans la production des protéines. L’ARN remplit d’autres fonctions relatives aux réactions chimiques du métabolisme cellulaire.
Les ribosomes sont des structures qui servent à fabriquer les protéines de la cellule. Ils se situent dans un compartiment nommé Réticulum endoplasmique.
Les mitochondries, sur lesquelles je reviendrai, sont le principal site de la respiration cellulaire. Elles assurent la production énergétique en dégradant sucres et lipides.
La vacuole maintient l’équilibre de l’intérieur de la cellule. Elle gère les bonnes concentrations des éléments dans le cytoplasme, en stockant sélectivement ces derniers au sein de sa membrane. Le ribosome est un complexe qui assure la traduction des ARNm (ARN messagers) en protéines. Pour cela, l’ARNm se charge de copier et transmettre l’information contenue dans le génome.
ADN nucléaire :
L’ADN (acide désoxyribonucléique), présent au sein du noyau de la cellule, contient les informations génétiques indispensables aux fonctions vitales de cette dernière. L’ensemble de ces informations constitue le génome qui porte toutes les données relatives à la morphologie et aux fonctions physiologiques transmises par hérédité.
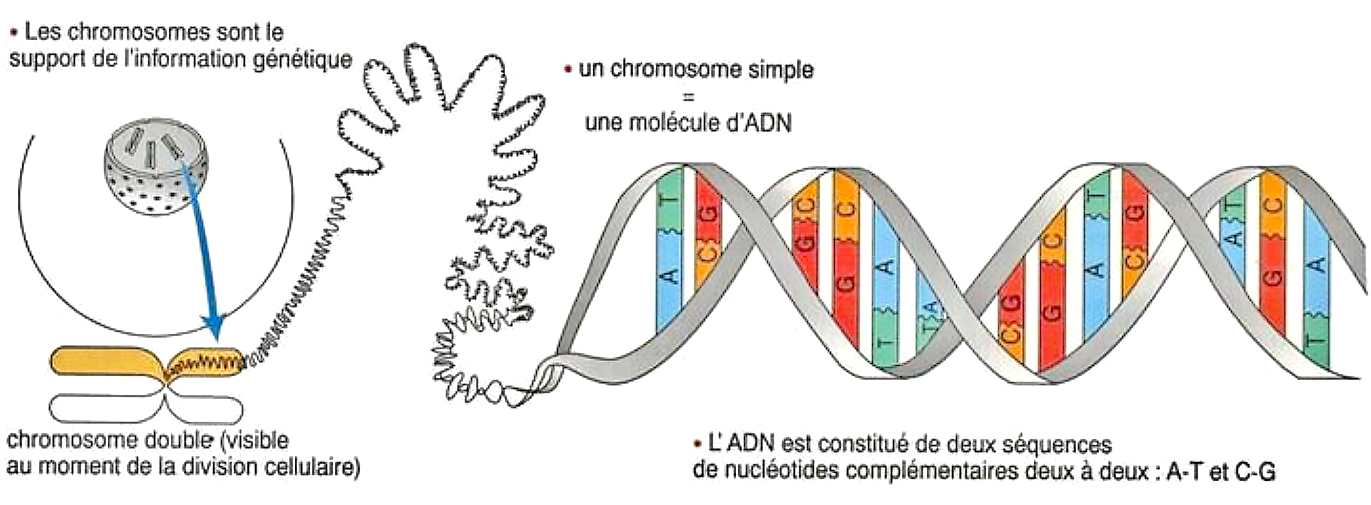
Ci-dessous, schéma de chromosomes constitués de protéines basiques associées à l’ADN (dit nucléaire) qui se présente sous la forme d’une macromolécule. Le chromosome porte les informations définissant des caractères. Par exemple, chez l’homme, la taille, la couleur des yeux, l’implantation des cheveux, le facteur de risque par rapport à certaines maladies, etc...
L’ADN nucléaire a une taille impressionnante qui dépasse un mètre de long une fois développé. En effet, il se compose d’un certain nombre de brins très fins, embobinés selon un agencement particulier, associés entre eux sous forme de paires constituées d’éléments nommés nucléotides (désoxyribonucléotides). Ces derniers qui constituent l'élément de base de l'ADN et de l'ARN sont identifiés par des lettres* A-T et G-C. Chez l’homme, on compte 46 brins, réunis en 22 paires, auxquelles s'ajoute une paire de chromosomes définissant le sexe (XX ou XY).
Les nucléotides sont au nombre de quatre : Le dAMP dont la base nucléique est l'adénine (A), le dGMP, dont la base nucléique est la guanine (G), le TMPa dont la base nucléique est la thymine (T) et le dCMP dont la base nucléique est la cytosine (C).
ADN mitochondrial :
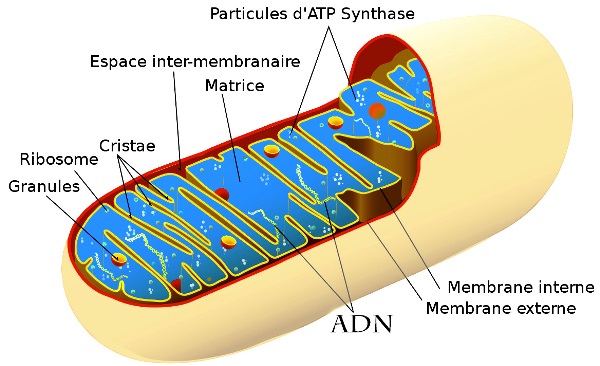 Il se présente sous la forme d’une molécule circulaire qui code essentiellement les protéines des ARN (ribosomaux et de transfert) spécifiques au fonctionnement de la mitochondrie.
Il se présente sous la forme d’une molécule circulaire qui code essentiellement les protéines des ARN (ribosomaux et de transfert) spécifiques au fonctionnement de la mitochondrie.
De taille inférieure à l'ADN nucléaire, Celui de l’homme porte 16.569 paires de nucléotides (l'ADN nucléaire en porte plus de 3.3 milliards).
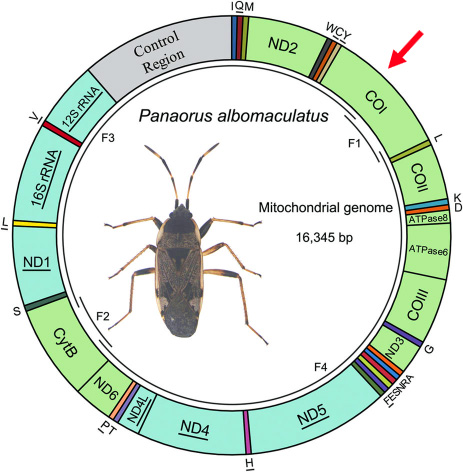
Ci-dessous, une carte du génome mitochondrial d’un hémiptère, sur laquelle on distingue des divisions qui correspondent chacune à un gène (un gène étant une portion du génome, autrement dit un segment de l'ADN). Dans le cas de cet insecte, treize de ces gènes sont codants pour la protéine (c'est le cas de COI que j'ai marqué d'une flèche rouge).
L'ADN des êtres vivants possède le même type de structure de base, soit : deux brins constitués chacun par une succession ordonnée de plusieurs milliers de nucléotides.
En revanche, selon les espèces, on peut constater des différences comme :
- Le nombre de molécules d’ADN dans une cellule.
- Sa longueur (quelques milliers à plusieurs milliards de nucléotides)
- Sa forme (linéaire ou circulaire)
- Sa localisation (noyau, mitochondrie ou cytoplasme)
Mais, c’est surtout la position des nucléotides (A, G, T, C) nommée "séquence" qui caractérise l’information génétique. Comme on va le voir, l’ADN mitochondrial est plus particulièrement utilisé pour le séquençage du génome en raison de ses propriétés intrinsèques dont, le fait qu’il est 5 à 10 fois plus spécifique que le génome nucléaire.
Remarque: Les régions d’ADN dites codantes, qui sont celles dont le code intervient pour la synthèse de protéines, se nomment les gènes, les autres régions de l’ADN sont dites non codantes (chez l’homme, ces dernières couvrent 98 % de l’ADN)
Taxonomie et code-barre ADN :
Malgré l’apparition des techniques d’identification génétiques sur la base du code-barre ADN, les biologistes définissent encore majoritairement les espèces à l’aide de clefs de détermination issues de l’observation de leurs caractéristiques communes, en partant des plus générales aux plus particulières. Ce travail nécessite la collecte de spécimens vivants ou suffisamment bien conservés pour permettre de lister de manière exhaustive leurs détails morphologiques. Le recours au code barre ADN est surtout en usage dans les laboratoires spécialisés dans la génétique. Les nouvelles espèces font donc prioritairement l'objet de descriptions morphologiques détaillées même si de plus en plus leur code barre vient compléter cette description.
Ci-dessous, photographie d’un collembole Deutonura sp. (sous-famille : Neanurinae).

Ci-après, je reprends un exemple de description de particularités morphologiques pour une nouvelle espèce de collembole Deutonura gibbosa (Porco, Bedos & Deharveng, 2010) appartenant à la sous-famille des Neanurinae, au sein de la famille des Neanuridea (Poduromorphe).
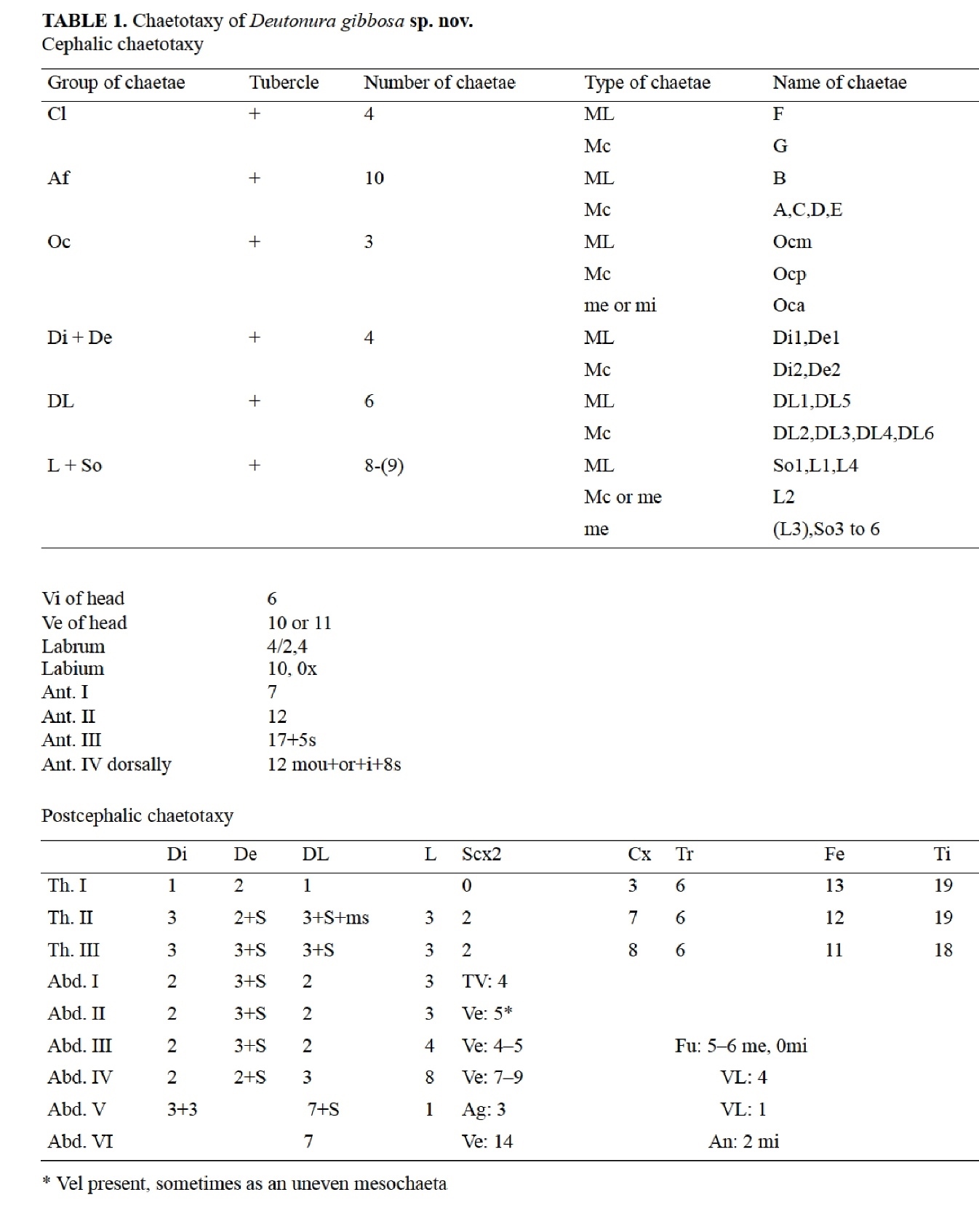
En taxonomie traditionnelle, chaque détail morphologique est répertorié sous forme d’un inventaire comprenant dessins et tableau de synthèse selon une terminologie abrégée spécifique.
Dans le cas présenté de Deutonura gibbosa sp. nov les poils sont identifiés selon :
- Leur nature : ML (macroseta long), MC (macroseta court), Me (mesosetae), Mi (microsetae)
- Leur implantation : Th.I (thorax, segments I), Abd.4 (abdomen, segment IV), Ant.2 (antenne, second segment) etc…
- Leur nombre.
Sur la planche suivante différentes parties du corps sont représentées pour répertorier l’implantation des poils : fig. 2 : Dos / fig. 3: Labrum / fig. 4: Labium / fig. 5: tibio-tarse et griffe de la jambe 1 / fig. 6 : ventre au niveau de l’abdomen. (Revoir articles « morphologie et physiologie »).

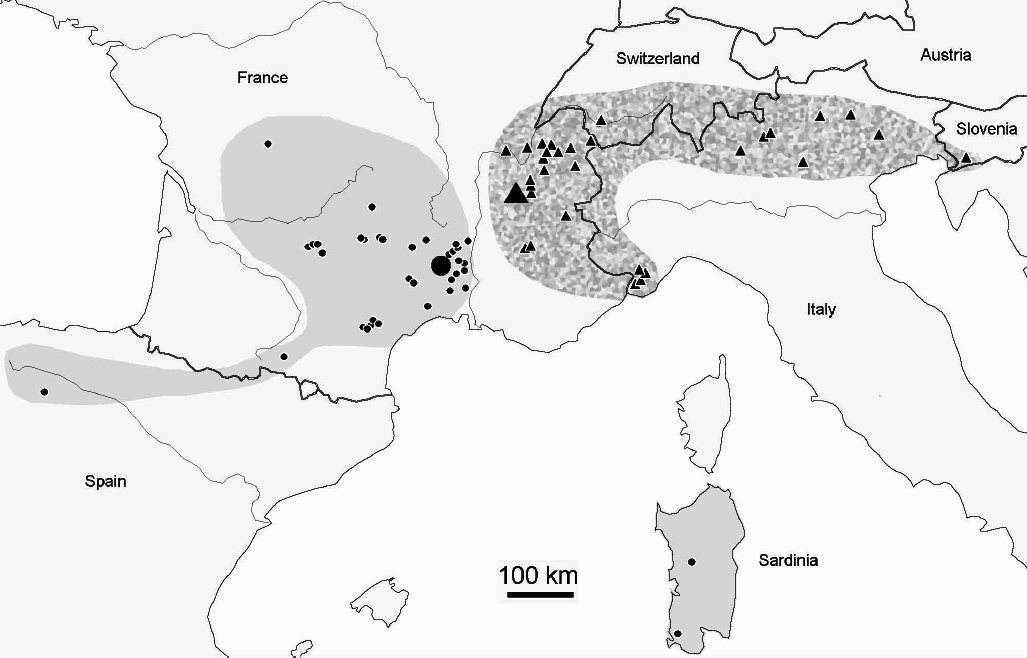
 On trouve également, pour accompagner ces descriptions, des cartes montrant les répartitions géographiques des espèces ainsi que la liste des lieux est le nombre de spécimens qui y sont prélevés.
On trouve également, pour accompagner ces descriptions, des cartes montrant les répartitions géographiques des espèces ainsi que la liste des lieux est le nombre de spécimens qui y sont prélevés.
Si documentées soient-elles, ces clefs d’identification demeurent pourtant incomplètes car elles ne permettent pas de mettre en évidence des diversités autres que morphologiques, on parle alors de diversités cryptiques*. En outre, le manque de ressources humaines dédiées au sein de la communauté scientifique ralentit considérablement l’avancement de ces travaux. A ce propos, on peut remarquer que les animaux invertébrés qui regroupent insectes, arachnides, collemboles, etc. sont d’une telle diversité qu’on estime aujourd'hui que 80% d’entre eux nous sont encore inconnus.
* Espèces cryptiques. L’individualisation génétique pour des spécimens indissociables au niveau morphologiques a été mise en évidence lors d’une étude (James et al., 2010) qui montre que la description du Lumbricus terrestris (ver de terre) s’appliquait en fait à deux espèces distinctes Lumbricus terrestris et Lumbricus herculeus. (Decaëns al., 2013) estiment que 30% en moyenne des espèces définies selon leur morphologie seraient des complexes d’espèces cryptiques. Une autre étude (Janzen et al., 2013) montre que 32 espèces de papillons Saturnidae décrites morphologiquement, représentent en réalité 49 espèces distinctes au plan génétique. Ces seuls exemples illustrent le nouveau risque que l’on nomme « chimère biologique » qui réduirait plusieurs espèces véritables à une dénomination unique.
Code-barre ADN :
Afin de pouvoir aller plus loin dans l’approche taxonomique, les chercheurs ont abordé cette question sur le plan de la génétique. A cette occasion, ils ont remarqué qu’il n’était pas nécessaire de séquencer la totalité de la chaîne d’ADN d’un spécimen pour pouvoir discriminer son espèce. En effet, Ils ont découvert que l’analyse de portions (séquences) de cette longue chaîne était suffisante (voir ci-dessous)
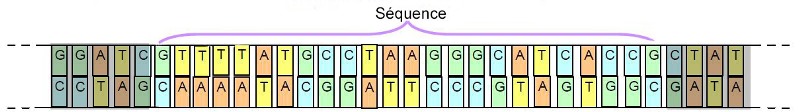
Important : Pour définir cette séquence, il faut cependant en identifier une qui varie d’une espèce à l’autre tout en demeurant stable pour une même espèce.
Depuis le milieu des années 1980, les bactériologues ont utilisé le séquençage génétique dans le cadre de leurs diagnostics des infections bactériennes (légionellose, certaines formes de méningite, etc..). Le gène séquencé étant l'ARN ribosomique 16S. Pour en savoir plus, cliquer ici (téléchargement PDF).
En 2003, Paul Herbert (Directeur du Biodiversity Institute of Ontario) et son équipe, axent leurs recherches sur le séquençage d'un gène (portion définie de l’ADN qui correspond à une unité d’hérédité de base) différent, provenant d’une région spécifique de l’ADN présent dans le génome mitochondrial des cellules eucaryotes. Cette portion d’ADN devenue un standard d’identification correspond à une partie du gène qui code la "sous-unité 1 de la cytochrome C Oxydase", une protéine nommée CO1 (situé dans la zone marquée d’une flèche rouge sur le second schéma du paragraphe "ADN mitochondrial" ) il constitue une portion du code d’une enzyme "Cytochrome c Oxydase" indispensable à la biochimie de la chaîne respiratoire.
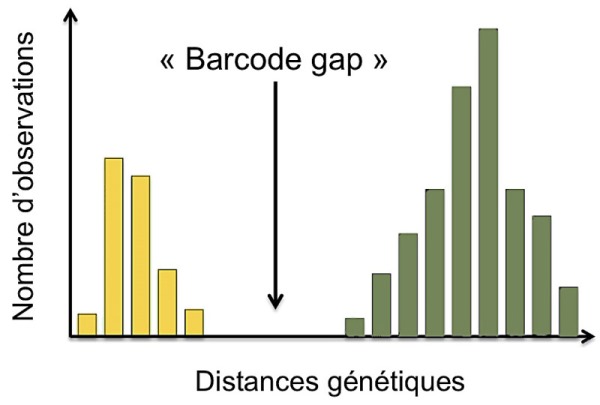 Définition : La distance génétique est un indice (valeur numérique) proportionnel au nombre de différences qui existent entre deux séquences d'un même fragment de gène et qui peut prendre en compte différents modèles d'évolution.
Définition : La distance génétique est un indice (valeur numérique) proportionnel au nombre de différences qui existent entre deux séquences d'un même fragment de gène et qui peut prendre en compte différents modèles d'évolution.
Par exemple, l’illustration ci-contre montre une différence significative des distances génétiques, en jaune pour des individus d’une même espèce (intra-spécifique) et en vert pour des individus appartenant à des espèces différentes (inter-spécifique).
Le gène CO1 choisi par Herber, tout en répondant a la nécessité première évoquée précédemment, présente aussi d’autres avantages, comme par exemple sa vitesse d’évolution élevée et le fait qu’il permet une accumulation appréciable de mutation au sein d’une espèce. Il apparaît, par comparaisons de séquences ADN, que le gène CO1 diverge suffisamment pour permettre une discrimination des espèces, voire la discrimination de populations au sein d’une même espèce (Cox and Hebert 2001- Wares and Cunningham 2001). En outre, comme il est déjà très présent au sein de la cellule qui compte entre 300 et 2000 mitochondries et également sous forme de copies (de 5 à 10) dans la mitochondrie elle-même, il est relativement facile à multiplier, par une technique biochimique complexe nommée « amplification par PCR » (Polymerase Chain Reaction). Ce procédé permet l’obtention un très grand nombre de copies (100 à 10000) d’une séquence donnée d’ADN afin de disposer d’une quantité suffisante pour permettre aux machines d’assurer le séquençage.
Outil d’investigation :
L’utilisation du code barre ADN apparaît donc aujourd'hui comme un outil qui élargit considérablement le champ d’investigation des biologistes. Alors que la taxonomie classique montre ses limites, en ce qu’elle nécessite d’étudier de spécimens intacts, l’étude génétique s’accommode de divers états comme par exemple les œufs ou les larves mais aussi des fragments de cadavres, d’excréments, de mucus ou d’autres traces présentes dans divers substrats (on parle à ce propos d’analyse d’ADN intracellulaire et d’analyse d’ADN extracellulaire). Ainsi, l’analyse de l’eau d’une marre peut révéler qu’une espèce donnée de grenouille y a évoluée, même si cette grenouille n’a pu y être physiquement prélevée.
Le Code-barre ADN ouvre également la voie à la reconstitution historique, tant au niveau des mécanismes de séparation de deux espèces que dans l’évolution de leur répartition géographique (phylogéographie). A ce propos, D. Porco et al., 2013 ont montré comment des collemboles européens introduits en Amérique du Nord y sont devenus invasifs.
Remarque : On trouve de nombreux cas*d’espèces introduites accidentellement, en particulier par le biais d’échanges commerciaux entre continents. Leur caractère est souvent invasif du fait de l’absence de prédateurs naturels, conduit parfois à envisager d’introduire ces derniers… non sans risques !
L’utilisation du code-barre ADN permet également de mettre en évidence les comportements prédateurs de certaines espèces à travers l’analyse de leurs régimes alimentaires, par prélèvement des contenus de tubes digestifs ou des déjections. Ces études donnent un nouvel éclairage sur les interactions proies/prédateurs ou hôtes/parasites.
Le code-barre ADN qui peut être reproduit selon les besoins permet des analyses massives avec un gain de temps considérable comparé à l’identification morphologique dont on a vu les limites. Il permet en outre d’effectuer ce travail à partir d’états peu ou pas décrits par la taxonomie traditionnelle.
Cette technique a cependant ses propres limites. Effectivement, les chercheurs ont parfois constaté des similitudes génétiques sur des spécimens morphologiquement distincts ou, pour des populations géographiquement isolées où une même espèce peut présenter une forte variabilité génétique. Pour conforter leurs conclusions, les scientifiques croisent donc les résultats du code-barre ADN avec d’autres données d’identification.
* par exemple: Leptoglossus occidentalis, tortue aux ouïes (qui menace la Cistude d’Europe dont elle concurrence l’habitat), écureuil gris (qui supplante notre écureuil roux), frelon asiatique (tueur d’abeilles), le moustique tigre, le poisson chat (Ictalurus melas) dans nos étangs, etc…
Le séquençage ADN:
La préparation du séquençage nécessite une succession d’étapes qui consiste en une destruction des tissus suivie d'une purification et d'une "amplification" réalisée sous forme d’un protocole qui régit une série d’opérations physico-chimiques :
- Extraction de l’ADN à partir d’un échantillon choisi prélevé sur l’organisme à étudier.
- Démarrage de l’amplification « PCR » et suivi des diverses réactions chimiques qui s’y opèrent.
- Purification par piégeage et exclusion des particules résiduelles susceptibles de perturber le séquençage.
- Isolement de la séquence du brin par électrophorèse capillaire basée sur une analyse spectrale de la fluorescence des quatre marqueurs (fluorochromes)
- Récupération de la séquence mitochondriale par mesure de la fluorescence sur un capteur CCD puis corrections (calibration spatiale et spectrale) et collecte des données sous forme informatique.

Réalisé en laboratoire par des machines automatisées, le séquençage s’effectue à partir d’un tube à essai contenant ADN, nucléotides et amorce de l’ADN polymérase (issus de l’amplification PCR évoquée plus haut).
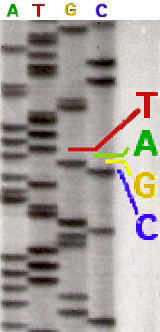 Je ne saurais détailler ici les complexes réactions de synthèse qui ont lieu dans ce tube. On peut juste noter que l’analyse spectrale n’est possible que grâce au marquage par des fluorochromes différents (A vert, T rouge, G jaune et C bleu), chaque couleur occupant des plages de longueurs d’onde distinctes. Lors de la restitution de l’analyse chaque nucléotide apparaît donc dans une succession correspondant à son ordre au sein de la séquence analysée.
Je ne saurais détailler ici les complexes réactions de synthèse qui ont lieu dans ce tube. On peut juste noter que l’analyse spectrale n’est possible que grâce au marquage par des fluorochromes différents (A vert, T rouge, G jaune et C bleu), chaque couleur occupant des plages de longueurs d’onde distinctes. Lors de la restitution de l’analyse chaque nucléotide apparaît donc dans une succession correspondant à son ordre au sein de la séquence analysée.
Le marquage radioactif qui fut initialement utilisé ne permettait pas de distinguer un nucléotide des trois autres. Il fallait alors séquencer à partir de quatre tubes différents, chacun possédant un type de nucléotide. Un séquençage avec marquage radioactif pouvait être lu directement à l’œil sur un gel d’acrylamide (ci-contre).
De nos jours, le remplacement du marquage radioactif par des fluorochromes autorise la réalisation des quatre réactions au sein d’un même tube. Les séquenceurs modernes détectent les intensités de fluorescence et possèdent des logiciels qui permettent de traduire les résultats sous forme graphique, comme on peut le voir dans l’exemple ci-dessous.
On remarque sur la partie supérieure, la succession des nucléotides repérés par leur lettre et accompagnées de chiffres qui permettent de les situer au sein de la séquence étudiée. Sur la partie inférieur, les courbes de quatre couleurs correspondent à l’amplitude (intensité) des fluorescences mesurées pour chacun des marqueurs.
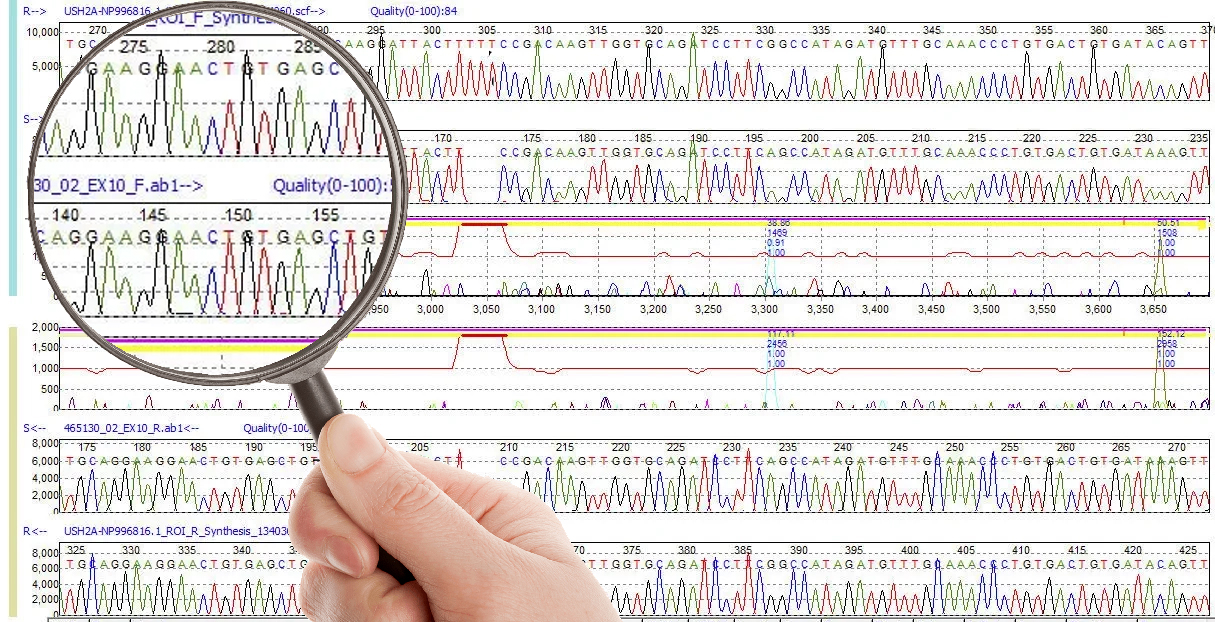
Au-delà de ce type de présentation, toutes les informations sont également consignées sous formes de tableaux utilisant une nomenclature internationale qui permet des comparaisons automatisées avec les bases de données existantes.
Bases de données :
Les bases de données génétiques deviennent cruciales en biologie, de ce fait la publication d’un article scientifique décrivant une séquence biologique est assujettie au dépôt préalable de ses données génétiques sur une des trois bases suivantes :
- Genbank (USA). Natural center for Biotechnology Information
- EMBL (Europe.) Nucleotide Sequence Database
- DDBJ (japon). Data Bank of Japan
Le nombre de séquence ou de génomes entiers qui y sont déposés s’est accru de manière exponentielle entre 2000 et 2014 alors que parallèlement les coûts des séquençages diminuaient dans des proportions similaires facilitant d’autant les recours à ce type de techniques.
Aujourd'hui, il existe des centaines de bases de données, ce qui ont conduit à la création d’une base de données des bases de données : NAR database
A titre indicatif, en septembre 2012 les bases de données nucléiques comptaient, pour les seuls invertébrés, 31.907.138 entrées, soit un total de 52.527.673.643 nucléotides !
Dans le cas spécifique des code-Barre ADN, il existe, par exemple, une base de données gérée par International Barcode of Life qui au 02/05/2018 disposait dans sa plateforme bio-informatique BOLD d’un ensemble de plus de 6 millions de codes-barre ADN dont, pour le règne animal, 4.367.942 codes-barres représentant 154.271 espèces et issus de 238 pays. Cette plateforme dispose en outre d’outils permettant de comparer la séquence ADN d’un organisme inconnu à l’ensembles des code-barre ADN répertoriés dans la base.
Exemple d’étude :
Poursuivons l’exemple de Deutonura gibbosa sp.nov (Porco-Bedos & Deharveng, 2010) mentionné précédemment pour lequel ces chercheurs ont réalisé une étude de code-barre ADN.
Le genre Deutonura qui compte plus de 56 espèces est un des principaux représentant de la grande sous-famille Neanurinea qui en compte au total 784 (dernière actualisation au 28-03-2018). Si certaines espèces comme Deutonura phleagea (Caroli, 1912- Cassagnau, 1979) possèdent une large aire de répartition, la majeure partie d’entre elles ont été observées dans des zones bien plus restreintes, certaines mêmes étant des espèces endémiques étroites. La répartition géographique de Deutonera gibbosa sp.nov (espèce très commune du genre) couvre les Alpes et le sud du Jura.
Observation :
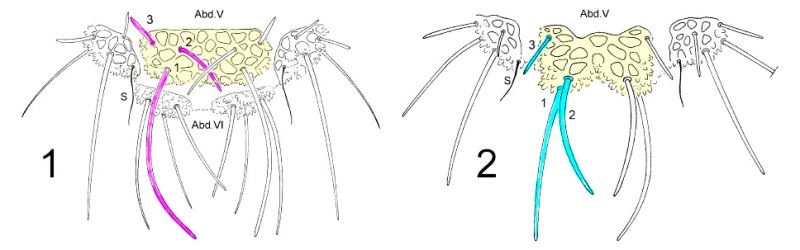
Pour dissocier les espèces du genre Deutonera, la taxonomie classique prend principalement en compte l’arrangement des tubercules dorsaux ainsi que le nombre de poils sur chaque tubercule. L’observation des deux espèces, Deutonura deficiens sylvatica (fig.1) et Deutonura gibbosa sp.nov. (fig.2) montre cependant une implantation similaire des poils dorsaux et un même nombre par tubercules.
La seule différence constatée, comme le montre le croquis ci-dessous, réside en une bilobation et un allongement du tubercule dorso-interne sur Abd.V (en jaune), associés à une modification de l’arrangement des poils et de leurs longueurs sur ce même tubercule.
Séquençage :
Les chercheurs ont effectué un séquençage sur 17 spécimens de 5 espèces du genre Deutonura (6 D. caerulescnens, 5 D. deficiens sylvatica, 1 D. decolorata, 4 D. gibbosa et 1 D.vallespirensis). Les distances génétiques mesurées sont reportées sur les tableaux ci-dessous : Tableau.1 (interspécifiques), Tableau.2 (intraspécifiques).

Ces valeurs sont également illustrées par l’arbre ci-dessous, généré à partir d’un modèle de transcription des nucléotides nommé « K2P » (Kimura deux paramètres) sur lequel les côtés supérieurs et inférieurs des triangles sombres font apparaître respectivement les distances génétiques maximales et minimales au sein des espèces.
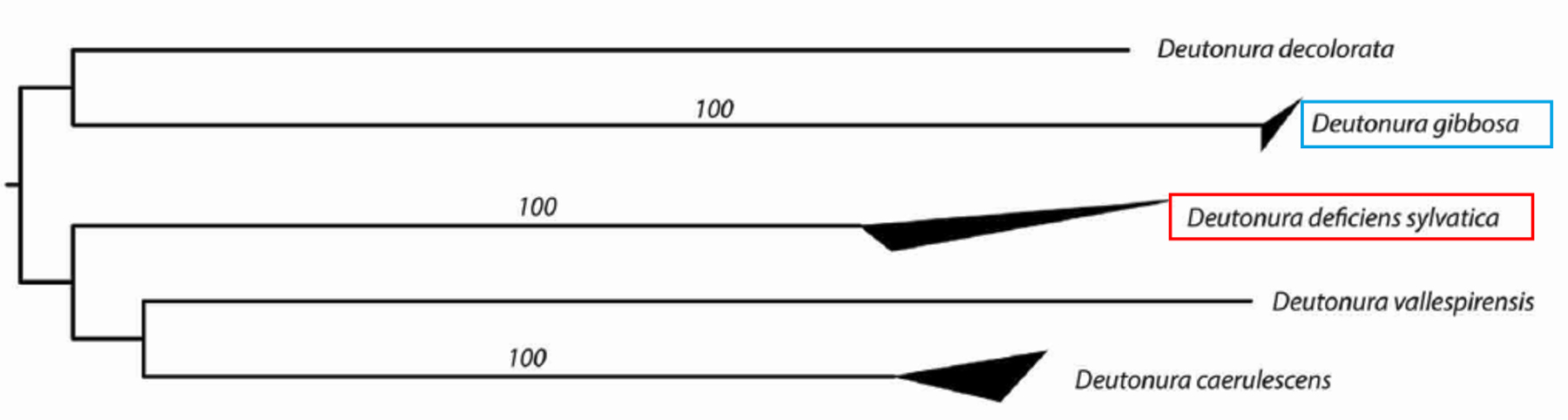
On visualise effectivement que Deutonura gibbosa (Barcoding effectuée avec 4 spécimens), possède la plus faible valeur intraspécifique (0.0006) du genre Deutonura alors que pour son plus proche voisin, Deutonura deficiens sylvatica, la mesure effectuée à partir des 5 spécimens, montre une distance interspécifique de 0,2074.
Ces deux valeurs intra et interspécifiques ainsi que les distances caractérisant les autres espèces de ce même genre (Deutonura) apportent un support génétique permettant de délimiter Deutonura gibbosa en tant que lignée mitochondriale distincte.
Conlusion :
Comme le souligne cet exemple, les différences morphologiques minimes entre deux espèces D. gibbosa et D. deficiens sylvatica sont nettement plus marquées sur le plan génétique. Ce constat effectué dès les années 2010 avec lequel convergent des études plus récentes confirme le potentiel de l’outil génétique en ce qui concerne la caractérisation des espèces. Plus largement, de très nombreuses espèces non encore décrites ou qui nécessitent des examens approfondis bénéficient de cet apport qui accroît fiabilité et précision. Mais les ressources du code-barre ADN s’étendent aussi bien au-delà de l’identification des espèces en ce qu'elles impactent de manière plus globale l’ensemble du champ de la biodiversité.
Remerciements : Je remercie vivement David Porco, auteur de l'étude sur Deutonura qui m'a ici servi d'exemple. Il a pris du temps pour relire cet article et me proposer des correctifs en vue de le rendre plus compréhensible.
Sources :
• "Description and DNA barcoding assessment of the new species Deutonura gibbosa (Collembola: Neanuridae: Neanurinae), a common springtail of Alps and Jura" (D. Porco, A. Bedos & L. Deharveng. 2010.)
• "Amplification PCR- Ifremer" : http://www.bibliomer.com/
• Société Française d’Ecologie et d’Evolution : Article : "Le barcoding ADN" (T.Decaëns, D.Porco t R.Rougerie).
Images :
• Cellule : https://www.encyclopedie-environnement.org/vivant/symbiose-evolution-lorigine-de-cellule-eucaryote/
• Mitochondrie : https://fr.wikipedia.org/wiki/Mitochondrie
• ADN nucléaire : ADN et Chromosomes par L. Guibaud
• ADN mitochondrial
• Graphique ADN modifié à partir de: https://softgenetics.com/products/mutation-surveyor/
• Deutonura sp. : Ph.Garcelon.